Forschende von FAU, Uni Göttingen und Uni Tübingen entschlüsseln molekulare Funktionsweise
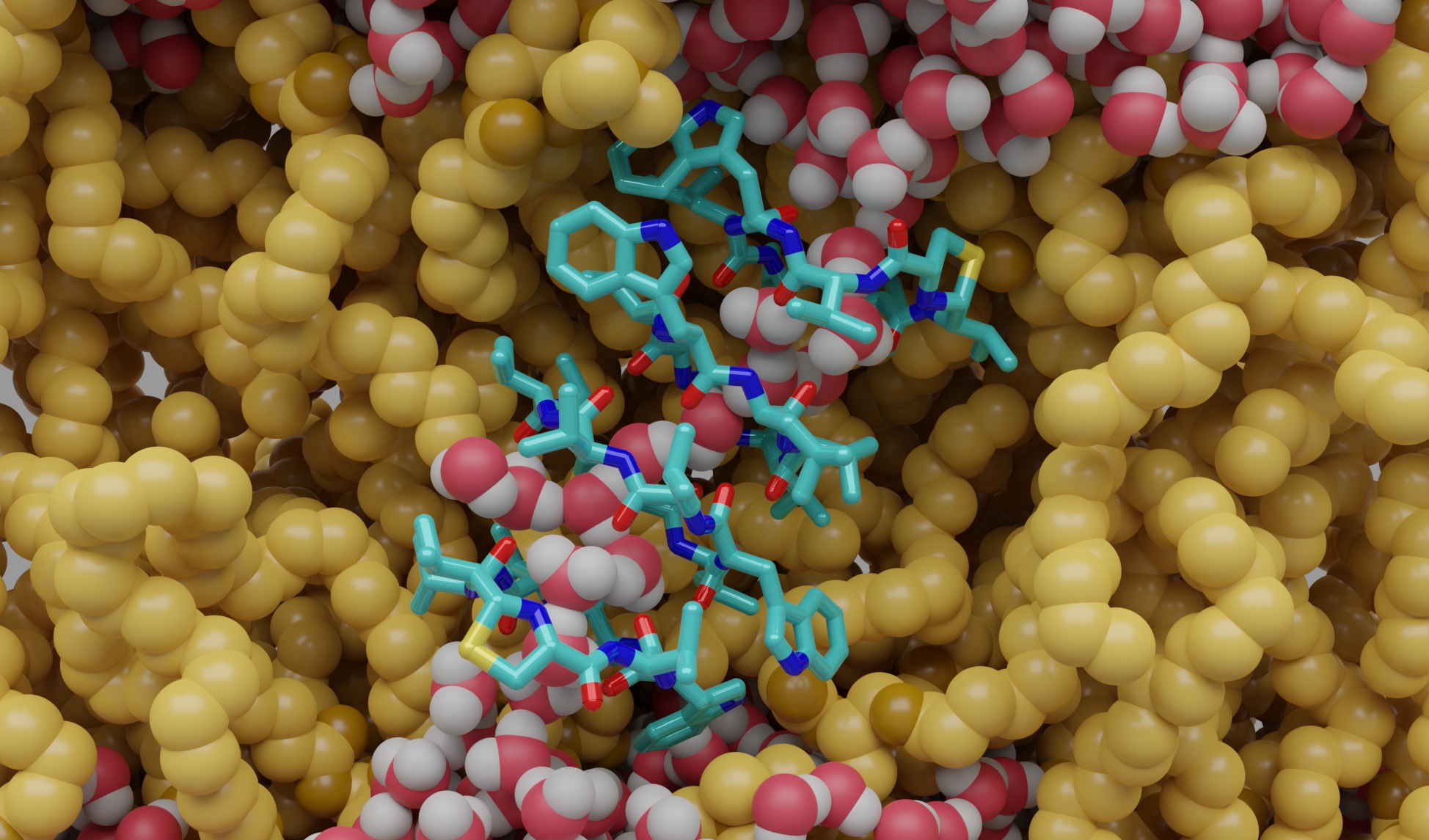
Auf der Haut oder in den Atemwegen des Menschen leben Milliarden von Mikroorganismen. Sie bilden das so genannte Mikrobiom. In der Beziehung zwischen diesen Mikroorganismen entstehen Stoffwechselprodukte, die für uns Menschen nützlich sind: Lugdunin etwa, ein zyklisches Peptid, das 2016 in einem Bakterium der menschlichen Nase entdeckt wurde und eine vielversprechende Wirkung gegen antibiotikaresistente Staphylococcus-Erreger gezeigt hat. Warum das so ist, war bislang unbekannt. Forschende der Friedrich-Alexander-Universität Erlangen-Nürnberg (FAU), der Uni Göttingen und der Uni Tübingen haben nun erstmals die Funktionsweise des Moleküls entschlüsselt.
Die genaue Wirkungsweise von kleinen Molekülen in Zellmembranen zu erforschen, ist sehr schwierig, da Membranen von Lebewesen hochkomplexe Systeme aus Millionen miteinander verbundener Bestandteile sind. Die Forschenden verwendeten daher vereinfachte Modelle dieser Membranen: Sie bestehen nur aus den einfachsten Bauteilen, den sogenannten Lipiden, und ermöglichen so eine gezielte Untersuchung der Interaktion von Lugdunin mit Zellmembranen ohne andere störende Faktoren. Um das Lugdunin in ausreichend großen Mengen zu erhalten, stellten die Forschenden es mittels chemischer Synthese her und führten dabei einige Veränderungen in die Struktur des Moleküls ein. Mit diesem Ansatz konnten sie herausfinden, dass sich einzelne Lugdunin-Moleküle in einer Membran zusammenschließen und aufeinanderstapeln. So entsteht eine hohle, röhrenförmige Struktur mit einem Durchmesser von weniger als einem Nanometer. Diese durchspannt die Membran und ermöglicht den Transport von Ionen. Mit einer speziellen Technik konnten sie den winzigen Ionenstrom über die Membran messen und damit belegen, dass Lugdunin auf diese Weise bakterielle Zellen bekämpft. (Video zur Bildung des Kanals, Video zum Transport von Wasser durch den Kanal)
Neben experimentellen Methoden verwendeten die Forschenden auch Computersimulationen, um ihre Ergebnisse zu untermauern.
Mehr Informationen: Doi: 10.1038/s41467-024-47803-6
Ansprechpartner:
Prof. Dr. Rainer A. Böckmann
Computational Biology – Theoretical & Computational Membrane Biophysics